走进XGBoost:强大且低成本的机器学习算法
看到Boost,很多朋友就想到了GBM(Gradient Boosting Machine)。是的,XGBoost和GBM都是应用梯度提升的算法,XGBoost的全称是eXtreme Gradient Boosting,是梯度提升最高效的应用。为什么这么有效?因为各种实际分析证明XGBoost是一个非常强大的分类和回归工具。尤其是在各种论坛比赛中证明了其非常强大,并且经常获胜。今天大海哥就带大家了解一下这个算法,让大家知道什么是
 head(diseaseInfo$ humans Affected)#转换前的列
head(diseaseInfo$ humans Affected)#转换前的列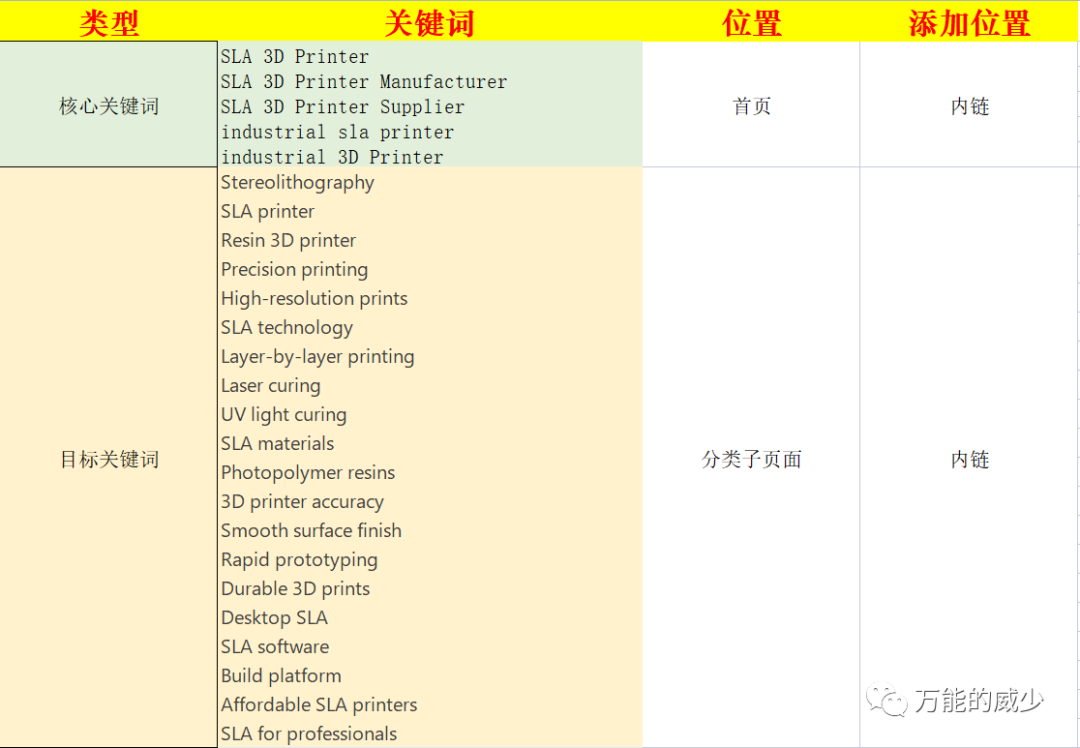
 #查看国家的属性
#查看国家的属性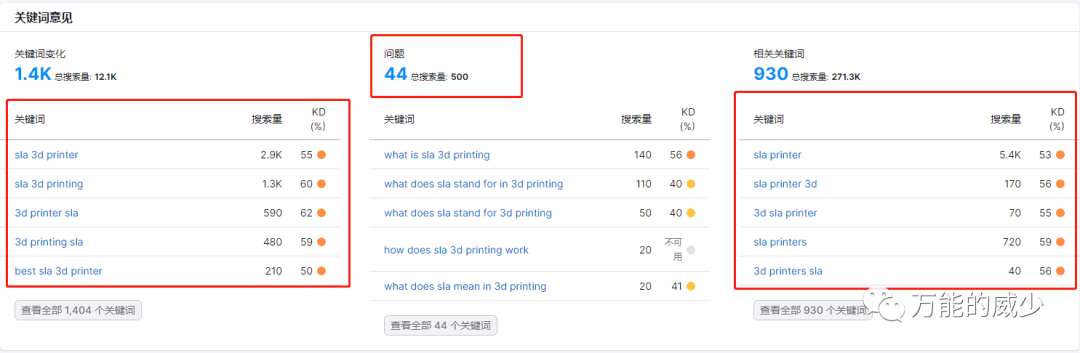
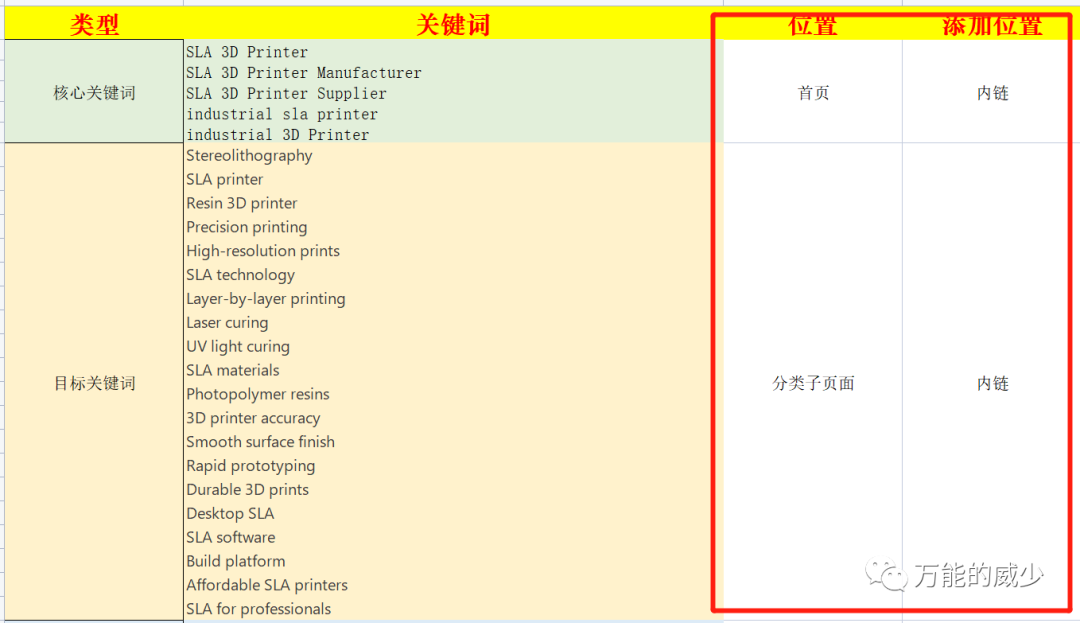
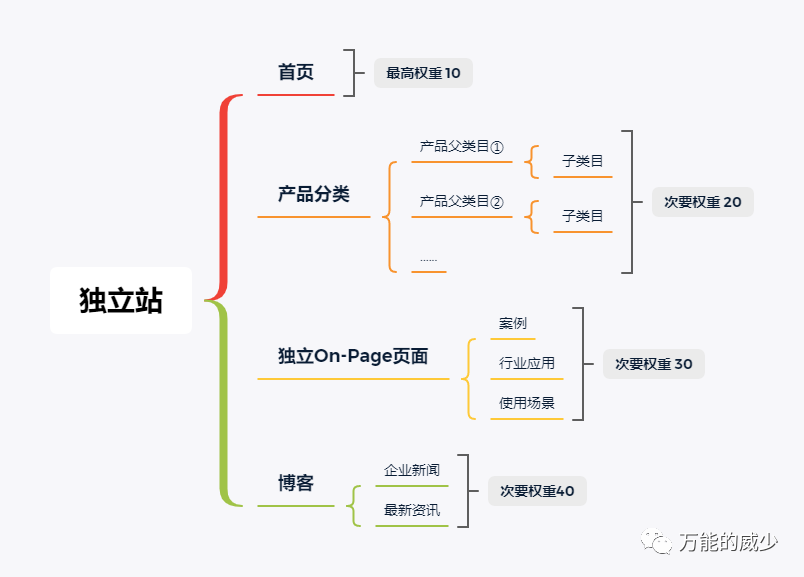

 基本上,这个故事告诉我们在查看组中的每棵树时每个特征的重要性。因此,增益较大的特征在我们的模型中非常重要,而增益较小的特征则没有多大帮助。
基本上,这个故事告诉我们在查看组中的每棵树时每个特征的重要性。因此,增益较大的特征在我们的模型中非常重要,而增益较小的特征则没有多大帮助。
XGBoost?
您应该如何准备数据?
如何使用XGBoost来训练和调整模型?
你的模型看起来和感觉如何?
首先我们要介绍一下,什么是XGBoost?
XGBoost 是梯度提升决策树算法的实现。那么什么是梯度提升决策树算法呢?我们通过下图来介绍一下。 ![]() 首先,我们创建新模型并将它们组合成组合模型字符串。该循环首先采用现有模型并计算数据中每个观察值的误差。然后我们建立一个新模型来预测这些错误。并将该错误预测模型的预测添加到“模型集合”中。不断迭代,每个错误都有一个对应的模型,然后将之前所有模型的预测相加。我们可以使用这些预测来评估新错误,构建下一个模型,并将其添加到组中。最后,我们的模型可以实现非常低的误差。其实这就是从0到1的过程,不断收集、收集、进步!大海哥还看到了一个比赛的结果,如下图。最终,XGBoost的表现还是相当恐怖的。仅经过 24 次训练,AUC 就达到了 0.9662。
首先,我们创建新模型并将它们组合成组合模型字符串。该循环首先采用现有模型并计算数据中每个观察值的误差。然后我们建立一个新模型来预测这些错误。并将该错误预测模型的预测添加到“模型集合”中。不断迭代,每个错误都有一个对应的模型,然后将之前所有模型的预测相加。我们可以使用这些预测来评估新错误,构建下一个模型,并将其添加到组中。最后,我们的模型可以实现非常低的误差。其实这就是从0到1的过程,不断收集、收集、进步!大海哥还看到了一个比赛的结果,如下图。最终,XGBoost的表现还是相当恐怖的。仅经过 24 次训练,AUC 就达到了 0.9662。 ![]() 介绍完规则,我们就开始分析吧!我们看看用哪个R包来分析
介绍完规则,我们就开始分析吧!我们看看用哪个R包来分析
library(xgboost)
library(tidyverse)
#主要就是这两个,是不是很方便,然后我们导入分析数据diseaseInfo <- read_csv("Outbreak_240817.csv")#然后开始处理一下数据集#划分一下训练集和测试集set.seed(1234)diseaseInfo <- diseaseInfo[sample(1:nrow(diseaseInfo)), ]#看看数据head(diseaseInfo)
#这个数据集为过去两年动物疾病爆发的时间、地点和内容,包括非洲猪瘟、口蹄疫和禽流感。病例数、死亡人数等也包括在内。本次分析我们想看看这些因素对人类是否有影响。# xgboost包要求数据有一定的格式,所以数据在开始分析之前需要做一下数据清理#首先删除一下人类相关的几列数据,大家也可以修改为自己想剔除的列,这里是为了教大家如何批量删除,同时刚好也删掉目标变量#删掉human开头的列diseaseInfo_humansRemoved <- diseaseInfo %>%select(-starts_with("human"))#还可以剔除掉一些冗余数据,同时删掉所有的非数字变量,部分分类变量转化为数值变量diseaseInfo_numeric <- diseaseInfo_humansRemoved %>%select(-Id) %>% select(-c(longitude, latitude)) %>%select_if(is.numeric)#把目标变量处理一下diseaseLabels <- diseaseInfo %>%select(humansAffected) %>% # 看看人类是否受影响这一列is.na() %>% # is it NA?magrittr::not() # 转换为TRUE和FALSEhead(diseaseLabels) #转换后的列
# 看看数据的格式str(diseaseInfo_numeric)
head (diseaseInfo$country)![]()
#我们需要把国家划分为几类,可以用one-hot编码处理一下model.matrix(~country-1,head(diseaseInfo))
#保留这个国家特征转化而来的的稀疏矩阵region <- model.matrix(~country-1,diseaseInfo)#有时,我们可能还需要进行一些文本处理。例如我们有一个speciesDescription特征,包含哪种特定动物患病以及是否是家养动物的信息,我们看一下
#这里主要有domestic和wild两个关键字,分别为家养和野生,那我们可以把他们转化为对应的两类diseaseInfo_numeric$is_domestic <- str_detect(diseaseInfo$speciesDescription, "domestic")#还可以根据物种描述绘制一个物种矩阵speciesList <- diseaseInfo$speciesDescription %>%str_replace("[[:punct:]]", "") %>% str_extract("[a-z]*$") # extract the least word in each row# 列表转换为矩阵speciesList <- tibble(species = speciesList)# 继续用one-hot编码转换一下options(na.action='na.pass') # 忽略NA值species <- model.matrix(~species-1,speciesList)#现在相当于我们有了三个数据diseaseInfo_numeric、region 和species,其实可以把他们捆绑起来diseaseInfo_numeric <- cbind(diseaseInfo_numeric, region, species)diseaseInfo_matrix <- data.matrix(diseaseInfo_numeric)#然后就可以开始划分测试数据和训练数据啦numberOfTrainingSamples <- round(length(diseaseLabels) * .7)# 训练集train_data <- diseaseInfo_matrix[1:numberOfTrainingSamples,]train_labels <- diseaseLabels[1:numberOfTrainingSamples]# 测试集test_data <- diseaseInfo_matrix[-(1:numberOfTrainingSamples),]test_labels <- diseaseLabels[-(1:numberOfTrainingSamples)]#然后转换为dmatrix,这是XGBoost的专属格式,也可以不转换,但是转换后训练更快哦!dtrain <- xgb.DMatrix(data = train_data, label= train_labels)dtest <- xgb.DMatrix(data = test_data, label= test_labels)#现在我们已经清理了测试和训练集并准备就绪,是时候开始训练我们的模型了。#那么人类相关的变量有好几个,预测哪一个呢,这里建议二分类是最好的。model <- xgboost(data = dtrain, # 数据nround = 2, #最大提升迭代次数objective = "binary:logistic")# binary:logistic代表二分类变量
#可以看到模型的输出,在第一轮和第二轮中,模型在训练数据上都有一定的误差。但是在减少,说明有进步!继续!#先看看预测怎么样pred <- predict(model, dtest)# 获取预测误差err <- mean(as.numeric(pred > 0.5) != test_labels)print(paste("test-error=", err))
#开始迭代!model_tuned <- xgboost(data = dtrain, # 数据max.depth = 3, #决策树最大深度nround = 2, #最大提升迭代次数objective = "binary:logistic") # 函数类型pred <- predict(model_tuned, dtest)# 获取误差err <- mean(as.numeric(pred > 0.5) != test_labels)print(paste("test-error=", err))
#好像没有提升,为什么?答案就是部分变量的尺度不同,而我们没有设置对应的权重,这里我们重新设置一下参数negative_cases <- sum(train_labels == FALSE)postive_cases <- sum(train_labels == TRUE)# 开始训练model_tuned <- xgboost(data = dtrain, max.depth = 3, #决策树最大深度nround = 10, #最大提升迭代次数#如果我们在这么多轮中没有看到改进,就停止early_stopping_rounds = 3, objective = "binary:logistic",scale_pos_weight = negative_cases/postive_cases) # 控制不平衡的类,设置权重# 为我们保留的测试数据生成预测pred <- predict(model_tuned, dtest)# 输出误差err <- mean(as.numeric(pred > 0.5) != test_labels)print(paste("test-error=", err))
#提升显著哦!#最后看一下哪些变量对于模型的贡献最大importance_matrix <- xgb.importance(names(diseaseInfo_matrix), model = model)xgb.plot.importance(importance_matrix)
版权声明
本文仅代表作者观点,不代表Code前端网立场。
本文系作者Code前端网发表,如需转载,请注明页面地址。
 code前端网
code前端网



